Inhaltsverzeichnis
Einleitung: Die Wichtigkeit der Proteinstabilität
Das Verständnis der Proteinstabilität ist für die moderne Biochemie, pharmazeutische Forschung und Biotechnologie von grundlegender Bedeutung. Die Proteinstabilität bestimmt nicht nur, wie lange ein therapeutisches Protein in Lösung aktiv bleiben kann, sondern auch seine Eignung für Formulierung, Lagerung und Transport (Wang 1999). Vom Enzym-Engineering über die Arzneimittelentwicklung bis hin zu strukturbiologischen Studien – die Fähigkeit, die Proteinstabilität genau zu bewerten und zu optimieren, ermöglicht es Forschern und Innovatoren, biowissenschaftliche Anwendungen voranzutreiben und gleichzeitig die Risiken aufgrund von Aggregation, Denaturierung oder Aktivitätsverlust zu minimieren.
Jüngste Veröffentlichungen unterstreichen die Rolle der Proteinstabilität bei der Formulierung von Arzneimitteln und bei der Bioprozessierung, wo selbst kleine Änderungen der Pufferzusammensetzung oder der Temperatur die Proteinfaltung und die therapeutischen Ergebnisse erheblich beeinflussen können (Durowoju et al. 2017).
Faktoren, die die Proteinstabilität beeinflussen
Die Funktion von Proteinen hängt entscheidend von der Unversehrtheit ihrer dreidimensionalen Struktur ab – der nativen Konformation. Diese Struktur kann durch eine Vielzahl von physikochemischen Faktoren gestört werden:
- Temperatur: Erhöhte Temperaturen können zur Entfaltung von Proteinen führen, was einen irreversiblen Funktionsverlust oder eine Aggregation zur Folge hat.
- pH-Wert: Änderungen des pH-Werts stören die ionischen Wechselwirkungen und Wasserstoffbrückenbindungen, was sich auf die strukturelle Stabilität auswirkt.
- Ionenstärke: Salze können Proteine je nach den spezifischen Wechselwirkungen mit geladenen Seitenketten stabilisieren oder destabilisieren.
- Ligandenbindung: Kleine Moleküle, Kofaktoren oder Medikamente, die an Proteine binden, können bestimmte Konformationen stabilisieren oder destabilisieren (Durowoju et al. 2017).
- Genetische Mutationen: Einzelne Punktmutationen können die Stabilität erhöhen oder verringern und damit das Verhalten des Proteins unter physiologischen Bedingungen beeinflussen.
Systematische Studien zeigen, dass die Anpassung der Pufferchemie und die Kontrolle der Temperatur während der Handhabung und Lagerung zu den wirksamsten Maßnahmen zur Erhaltung der Proteinaktivität gehören (Wang 1999).
Grundsätzliches: Was bedeutet Proteinstabilität?
Proteinstabilität bezieht sich auf die Widerstandsfähigkeit der nativen Struktur eines Proteins unter verschiedenen Bedingungen. Sie umfasst Folgendes:
- Thermodynamische Stabilität: Die Tendenz eines Proteins, gefaltet zu bleiben und nicht zu denaturieren.
- Kinetische Stabilität: Die Geschwindigkeit, mit der ein Protein in einen ungefalteten oder aggregierten Zustand übergeht.
- Umkehrbare Veränderungen: Einige Faltungsübergänge können durch Wiederherstellung günstiger Bedingungen rückgängig gemacht werden (z. B. Rückfaltung nach leichter Denaturierung).
- Unumkehrbare Veränderungen: Starke Verarbeitung (z.B. hohe Temperaturen oder chemische Mittel) kann zu einer Aggregation oder Entfaltung führen, die nicht rückgängig gemacht werden kann.
Die Stabilität wird häufig anhand der Schmelztemperatur (Tm) quantifiziert, bei der die Hälfte der Proteinpopulation denaturiert ist – ein Schlüsselparameter, der in Stabilitätsassays gemessen wird (Durowoju et al. 2017).
Methoden zur Bewertung der Proteinstabilität
Es wurden mehrere orthogonale Techniken entwickelt, um biomolekulare Stabilität und Wechselwirkungen zu untersuchen. Die Auswahl der geeigneten Methode hängt von der Forschungsfrage, dem Proteintyp und der verfügbaren Ausrüstung ab.
Dynamische Differenzkalorimetrie (DSC)
DSC ist ein unverzichtbares Werkzeug für präzise Stabilitätsstudien bei Proteinen (Durowoju et al. 2017). Durch die Messung der Wärme, die zur Entfaltung einer Proteinprobe bei steigender Temperatur erforderlich ist, bestimmt die DSC:
- Denaturierungstemperatur (Tm): Gibt die thermische Stabilität an.
- Enthalpie-Änderungen(ΔH): Zeigt Faltungsenergieübergänge an.
- Entfaltungsprofile: Charakterisieren Sie reversible oder irreversible Übergänge.
DSC wird für ihre Genauigkeit bei der Erkennung subtiler Übergänge – selbst bei Proteinen in niedrigen Konzentrationen – geschätzt und ist besonders leistungsfähig bei der Charakterisierung von Varianten, thermischem Formulierungsverhalten und ligandeninduzierten Stabilitätsverschiebungen (Qualification of a DSC Method 2020).
Andere Analysemethoden
Dynamische Lichtstreuung (DLS): Misst den hydrodynamischen Radius und erkennt die Proteinaggregation, ergänzend zur thermische Stabilität Assays (Mirasol et al. 2021).
UV-Vis-Spektroskopie: Überwacht die Entfaltung, indem Änderungen der Absorption in Verbindung mit aromatischen Seitenketten oder Chromophoren verfolgt werden.
Isotherme Titrationskalorimetrie (ITC): Quantifiziert Bindungsinteraktionen zwischen Proteinen und Liganden; nützlich für den Nachweis von Stabilitätsänderungen bei Ligandenassoziation (Durowoju et al. 2017).
Zirkulardichroismus (CD) Spektroskopie: Sondiert Veränderungen der Sekundärstruktur während der Faltung oder Denaturierung (z. B. Übergänge von Alpha-Helix zu Beta-Faltblatt).
Untersuchung von Wechselwirkungen, die die Stabilität beeinflussen
Proteine interagieren ständig mit ihrer molekularen Umgebung. Die Beurteilung, wie sich diese Wechselwirkungen auf die Stabilität auswirken, ist für Forschung und Entwicklung unerlässlich:
- Ligandenbindung: DSC und ITC werden verwendet, um die Bindungsaffinitäten und die stabilisierenden oder destabilisierenden Effekte von kleinen Molekülen, Medikamenten und Kofaktoren zu bestimmen (Durowoju et al. 2017)
- Protein-Aggregation: DLS identifiziert Veränderungen in der Aggregationsneigung während Hitze- oder Stresseinwirkung (Wang 1999).
- pH-Abhängigkeit: Durch die Durchführung von DSC-Läufen bei verschiedenen pH-Werten erhalten die Forscher Einblicke in die Rolle der Protonierungszustände bei der Faltung.
- Genetische Varianten: Der Vergleich von Tm- oder Aggregationsprofilen mittels DSC und DLS ermöglicht die Bewertung der Stabilität, die durch Punktmutationen oder konstruierte Sequenzänderungen entsteht (Mirasol et al. 2021).
Wie thermische Methoden die molekularen Wechselwirkungen beleuchten
Thermische Techniken wie DSC liefern nicht nur Tm und Enthalpie, sondern bieten in Kombination mit Liganden- oder Puffertitrierungsprotokollen auch tiefe Einblicke in die Bindungsaffinitäten und die strukturelle Stabilisierung (Durowoju et al. 2017). Ein Ligand, der die Tm eines Proteins erhöht, deutet zum Beispiel auf eine direkte stabilisierende Wechselwirkung hin, die für das Design oder die Formulierung eines Medikaments relevant ist.
Praktische Einblicke und Messparameter
Die Interpretation von Proteinstabilitätsdaten erfordert die Beachtung von Versuchsbedingungen und typischen Ergebnissen:
- Erhitzungsrate: Beeinflusst die Erkennung der kinetischen gegenüber der thermodynamischen Stabilität; langsamere Raten begünstigen Gleichgewichtsmessungen.
- Probenkonzentration: Eine ausreichende Konzentration gewährleistet ein zuverlässiges Signal ohne Aggregationsstörungen.
- Pufferzusammensetzung: Vergleichen Sie die stabilisierenden Effekte verschiedener Puffer und Hilfsstoffe auf Tm und Entfaltungsenthalpie (Wang 1999).
- Kontrollproben: Vergleichen Sie immer mit genau definierten Standards, z. B. Wildtyp vs. mutierte Proteine oder reine Pufferkontrollen.
Anwendung
Anwendungsbeispiele
- Vergleich der Stabilitätsprofile von monoklonalen Antikörpern für die Formulierungsentwicklung (Qualifizierung eines DSC Methode 2020).
- Evaluierung von Enzymvarianten für die industrielle Biokatalyse, die thermische Belastbarkeit erfordern.
- Screening von Protein-Ligand-Bindungsaffinitäten in der frühen Phase der Arzneimittelentdeckung und Strukturbiologie (Durowoju et al. 2017).
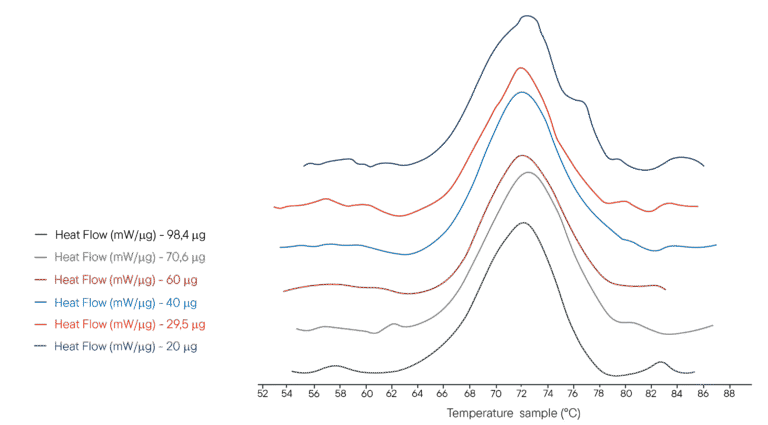
Anwendungsbeispiel: Thermische Denaturierung von Lysozym
100µl verschiedener Lösungen von Lysozym in PHB-Puffer wurden in einen 100µl-Tiegel gegeben. Es wurden verschiedene Konzentrationen verwendet, die jeweils unterschiedliche Massen des analysierten Lysozyms repräsentieren. Der Referenztiegel wurde mit dem gleichen Volumen an PHB-Puffer gefüllt. Die Ultimate DSC wurde so programmiert, dass sie einen Temperaturanstieg von 40°C auf 95°C bei 1°C/min durchführt.
Mit der Ultimate DSC können die Enthalpie und die Denaturierungstemperatur von Lysozym mit 5 bis 10 weniger Proben als mit den derzeit verfügbaren Geräten gemessen werden. Jedes Thermogramm wurde in weniger als 60 Minuten erstellt, was eine schnelle Analyse ermöglicht. Die Ultimate DSC spart große Mengen an Proben und ermöglicht es Ihnen, schneller zu arbeiten.
Wählen Sie die richtige Methode für Ihre Forschung
Die Wahl der optimalen Analysemethode hängt von Ihrem spezifischen Protein, Ihrem Forschungsziel und den verfügbaren Ressourcen ab:
| Method | Key Insights | Typical Applications |
|---|---|---|
| DSC | Tm, ΔH, unfolding profile | Formulation, variant screening, stability assays |
| DLS | Aggregation, size distribution | Formulation, quality control, aggregation analysis |
| ITC | Binding affinity, thermodynamics | Drug design, ligand screening, protein-protein interactions |
| CD-Spectroscopy | Secondary structure change | Conformational studies, denaturation monitoring |
| UV/Vis Spectroscopy | Folding/unfolding, aggregation | Quick screening, routine assay |
Für einen detaillierten Vergleich wird in aktuellen Übersichtsarbeiten hervorgehoben, wie die Integration mehrerer Techniken, einschließlich präziser kalorimetrischer und spektroskopischer Messungen, zu einer robusten Bewertung der Proteinstabilität und der Eignung für Downstream-Anwendungen führt (Mirasol et al. 2021; Durowoju et al. 2017).
Fazit & Nächste Schritte
Das Verständnis und die Kontrolle der Proteinstabilität sind von zentraler Bedeutung für eine erfolgreiche Biomolekülforschung, pharmazeutische Formulierung und Arzneimittelentwicklung. Die thermische Analyse, insbesondere die DSC, bietet robuste, quantifizierbare Einblicke in das Verhalten von Proteinen unter praktischen Bedingungen und hilft Ihnen dabei, datengestützte Entscheidungen mit Zuversicht zu treffen.
Wenn Sie Ihre Untersuchungen zur Proteinstabilität vorantreiben wollen, ist das Linseis UDSC L64 bietet modernste Kalorimetrie Möglichkeiten, die auf die biomolekulare Analyse und die Formulierungsentwicklung zugeschnitten sind. Erfahren Sie mehr über die Leistung und das vielseitige Anwendungsspektrum des UDSC L64 auf unserer Seite Kalorimetrie-Lösungen, oder wenden Sie sich an unser spezialisiertes Team für eine individuelle Beratung zu Ihren Forschungsanforderungen.
Ausgewählte Literatur zur weiteren Lektüre
- Durowoju, I.B., Bhandal, K.S., Hu, J., Carpick, B. und Kirkitadze, M. (2017) Differential Scanning Calorimetry-A Method for Assessing the Thermal Stability of Proteins, Journal of Visualized Experiments, (121), e55262. https://pmc.ncbi.nlm.nih.gov/articles/PMC5409303/
- Wang, W. (1999) ‚Instability, stabilization, and formulation of liquid protein pharmaceuticals‘, International Journal of Pharmaceutics, 185(2), pp. 129-188. https://doi.org/10.1016/S0378-5173(99)00152-0
- Mirasol, F., Wypych, J. und Kopec, B. (2021) Stability Testing of Protein Therapeutics Using DLS, Pharmaceutical Technology. https://www.pharmtech.com/view/stability-testing-of-protein-therapeutics-using-dls
- Qualifizierung einer Differential Scanning Calorimetry Methode für die biophysikalische Charakterisierung von monoklonalen Antikörpern (Open Access). https://www.openaccessjournals.com/articles/qualification-of-a-differential-scanning-calorimetry-method-for-biophysical-characterization-of-monoclonal-antibodies-an.pdf